论文:Reducing Snapshots to Points: A Visual Analytics Approach to Dynamic Network Exploration
作者:Stef van den Elzen, Danny Holten, Jorik Blaas, Jarke J. van Wijk
发表会议:VAST 2015
动态网络的可视化方法可以分为Animation (time-to-time) 和 Small-multiple (time-to-space) 两类。对于前者,用户每一时刻只能看到一帧,前后内容需要用户去记忆、理解,往往需要来回观看很多遍才能粗略理解动态网络的变化过程,给用户带来了很大的认知负担;对于后者,将不同时刻的网络进行并排罗列,由于屏幕空间有限,很多时候并不能将所有时刻的网络同时进行展示,且难以进行网络动态变化模式的发现。本文提出了一种对动态网络进行二维投影的方法,可以将每个动态网络表示成一张静态的node-link图,每个node代表某个时刻的网络,link连接了相邻时刻的node,可以有效地帮助用户发现稳定状态(stable state)、重现状态(recurring state)、异常拓扑(outlier topologies)及分析网络状态间的转移过程。
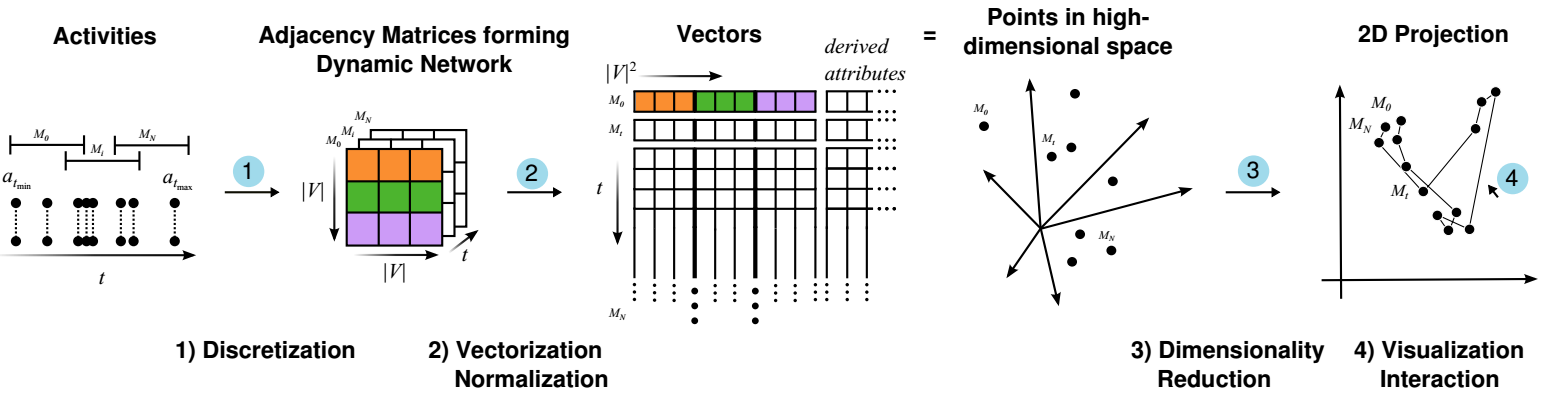
下图是本文方法的工作流程:
1. 离散化:原始数据由一系列时序的事件组成,每个事件均发生在某两个个体之间。方法的第一步是将原始数据进行离散化,生成一些离散的snapshots。具体做法是生成一些连续的时间窗(为了最后投影结果的连续性,时间窗口可以有部分重合),将每个时间窗内的所有事件转换成一个等价的网络,网络里的节点和边分别是这个时间窗内出现过的节点和边,并将每条边在这个时间窗内出现的次数当做该边的权重。
2. 向量化:为了进行投影,需要进行向量化将每个snapshot转换成一个高维的向量。具体做法是先将snapshot表示成邻接矩阵,然后将邻接矩阵转化成一个高维向量,当然这里还可以往该向量里加入一些额外的属性,比如该网络中节点平均的度(degree)等。
3. 投影:经过前面两个步骤,我们已经得到了一些列时序的高维向量,只需要对其进行二维投影即可。
继续阅读 =>


